NibabelとMayaviによるMR画像の可視化
PythonでNifti (The Neuroimaging Informatics Technology Initiative) 形式のMR画像の可視化をしてみる。まずNifti (nii.gz)形式のファイルはNibabelでloadする。Nibabelはpipで入る:pip install nibabel
なお、Nibabelに依存した機械学習ライブラリとしてNilearnというのもある。今回は使わない。
Nibabel tutorial
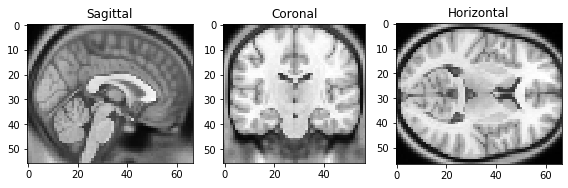
NibabelのtutorialにはEPI法(echo planar imaging)で撮影された拡散強調像 (someones_epi.nii.gz) と、(恐らく)T1 強調像 (someones_anatomy.nii.gz) の2つのデータが用意されている。どちらでもよいが、someones_anatomy.nii.gzの方をダウンロードしておく。
nibabelの関数(nib.load)でデータを読み込み、matplotlibで可視化する。
import nibabel as nib
import numpy as np
import matplotlib.pyplot as plt
from matplotlib import gridspec
# Data load
img = nib.load('someones_anatomy.nii.gz')
img_data = img.get_fdata()
print(img_data.shape) # (57, 67, 56)
# Plot
gs = gridspec.GridSpec(1, 3, width_ratios=[1.17, 1, 1.17])
plt.figure(figsize=(8,4))
plt.subplot(gs[0])
plt.title("Sagittal")
plt.imshow(np.flipud(img_data[28].T), cmap="gray")
plt.subplot(gs[1])
plt.title("Coronal")
plt.imshow(np.flipud(img_data[:, 33].T), cmap="gray")
plt.subplot(gs[2])
plt.title("Horizontal")
plt.imshow(img_data[:, :, 28], cmap="gray")
plt.tight_layout()
plt.show()
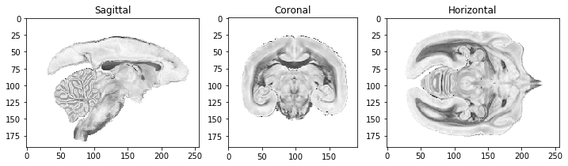
マーモセット脳MRIデータセット
マーモセット(marmoset)脳のMRIデータセット (Marmoset Brain Mapping)が公開された[1, 2]ので、それを読み込んで可視化してみる。
まず、dataのページから150um dMRIの前処理後のデータ(DTI-Fitted)をダウンロードする。これを選んでいるのは単にデータサイズが小さいため。このデータはマーモセットから脳を取り出した後、ホルマリン固定後、gadoliniumに浸して造影し、7T MRIでスキャンすることで得られたものである。
zipファイルを解凍後、DTIFIT_FA.nii.gzというデータ (添え字の詳細が無かった)を読み込んで可視化する。
Nibabelによるマーモセット脳MRIの断面図の可視化
コード自体は先ほどのtutorialのものとほぼ同じ。
import nibabel as nib
import numpy as np
import matplotlib.pyplot as plt
from matplotlib import gridspec
# Data load
img = nib.load('DTIFIT_FA.nii.gz')
img_data = img.get_fdata()
print(img_data.shape) # (192, 256, 192)
# Plot
gs = gridspec.GridSpec(1, 3, width_ratios=[1.33, 1, 1.33])
plt.figure(figsize=(10,4))
plt.subplot(gs[0])
plt.title("Sagittal")
plt.imshow(np.flipud(img_data[96].T), cmap="gray_r")
plt.subplot(gs[1])
plt.title("Coronal")
plt.imshow(np.flipud(img_data[:, 128].T), cmap="gray_r")
plt.subplot(gs[2])
plt.title("Horizontal")
plt.imshow(img_data[:, :, 96], cmap="gray_r")
plt.tight_layout()
plt.show()
Mayaviによるマーモセット脳の3Dでの可視化
Pythonで3次元Plotをするのにはmatplotlibやplotlyなどもあるが、今回はMayaviを用いる。installはpip install mayaviでできるが、
$ git clone https://github.com/enthought/mayavi.git
$ cd mayavi
$ pip install -r requirements.txt
$ pip install PyQt5 # replace this with any supported toolkit
$ python setup.py install # or develop
をすると確実。
import nibabel as nib
from mayavi import mlab
img = nib.load('DTIFIT_FA.nii.gz')
img_data = img.get_fdata()
fig = mlab.figure(size=(400, 400), bgcolor = (1,1,1))
src = mlab.pipeline.scalar_field(img_data)
mlab.pipeline.iso_surface(src,
contours=[img_data.min()+0.05*img_data.ptp(), ],
opacity=1,
color=(.5, .5, .5))
mlab.show()

実際にはグリグリと3Dで脳を動かしてみることができる。なお、脳溝がほぼ見えないが、マーモセットは元々脳溝が少ない。
参考文献
- Liu, C., Ye, F.Q., Newman, J.D. et al. A resource for the detailed 3D mapping of white matter pathways in the marmoset brain. Nat Neurosci. (2020) doi:10.1038/s41593-019-0575-0
- Liu, C., Ye, F. Q., Yen, C. C., Newman, J. D. et al. A digital 3D atlas of the marmoset brain based on multi-modal MRI. NeuroImage. (2018). doi:10.1016/j.neuroimage.2017.12.004